Il picco di SARS-CoV-2 compromette la riparazione del danno al DNA e inibisce la ricombinazione V(D)J in vitro
Dipartimento di Bioscienze Molecolari, The Wenner–Gren Institute, Università di Stoccolma, SE-10691 Stoccolma, Svezia
2
Dipartimento di microbiologia clinica, virologia, Università di Umeå, SE-90185 Umeå, Svezia
*
Autori a cui indirizzare la corrispondenza.
Redattore accademico: Oliver Schildgen
Ricevuto: 20 agosto 2021 / Revisione: 8 settembre 2021 / Accettato: 8 ottobre 2021 / Pubblicato: 13 ottobre 2021
La sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) ha portato alla pandemia della malattia da coronavirus 2019 (COVID-19), colpendo gravemente la salute pubblica e l'economia globale. L'immunità adattativa svolge un ruolo cruciale nella lotta contro l'infezione da SARS-CoV-2 e influenza direttamente gli esiti clinici dei pazienti. Gli studi clinici hanno indicato che i pazienti con COVID-19 grave mostrano risposte immunitarie adattive ritardate e deboli; tuttavia, il meccanismo con cui SARS-CoV-2 impedisce l'immunità adattativa rimane poco chiaro. Qui, utilizzando una linea cellulare in vitro, riportiamo che la proteina spike SARS-CoV-2 inibisce significativamente la riparazione del danno al DNA, che è necessaria per un'efficace ricombinazione V (D) J nell'immunità adattativa. meccanicamente, abbiamo scoperto che la proteina spike si localizza nel nucleo e inibisce la riparazione del danno al DNA impedendo il reclutamento della proteina chiave di riparazione del DNA BRCA1 e 53BP1 nel sito del danno. I nostri risultati rivelano un potenziale meccanismo molecolare mediante il quale la proteina spike potrebbe impedire l'immunità adattativa e sottolineare i potenziali effetti collaterali dei vaccini a lunghezza intera basati su spike.
Parole chiave: SARS–CoV–2 ; punta ; riparazione del danno al DNA ; ricombinazione V(D)J ; vaccino
1. Introduzione
La sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) è responsabile della pandemia di coronavirus 2019 (COVID-19) in corso che ha provocato oltre 2,3 milioni di morti. SARS-CoV-2 è un virus a RNA a singolo senso positivo con involucro che consiste di proteine strutturali e non strutturali [ 1 ]. Dopo l'infezione, queste proteine virali dirottano e disregolano il macchinario cellulare ospite per replicare, assemblare e diffondere virus della progenie [ 2 ]. Recenti studi clinici hanno dimostrato che l'infezione da SARS-CoV-2 influisce in modo straordinario sul numero e sulla funzione dei linfociti [ 3 , 4 , 5 , 6]. Rispetto ai sopravvissuti lievi e moderati, i pazienti con COVID-19 grave manifestano un numero significativamente inferiore di cellule T totali, cellule T helper e cellule T soppressorie [ 3 , 4 ]. Inoltre, COVID-19 ritarda i livelli di IgG e IgM dopo l'insorgenza dei sintomi [ 5 , 6 ]. Nel loro insieme, queste osservazioni cliniche suggeriscono che SARS-CoV-2 colpisce il sistema immunitario adattativo. Tuttavia, il meccanismo con cui SARS-CoV-2 sopprime l'immunità adattativa rimane poco chiaro.
Essendo due sistemi critici di sorveglianza dell'ospite, i sistemi immunitario e di riparazione del DNA sono i sistemi primari su cui si affidano gli organismi superiori per la difesa contro diverse minacce e l'omeostasi dei tessuti. Prove emergenti indicano che questi due sistemi sono interdipendenti, specialmente durante lo sviluppo e la maturazione dei linfociti [ 7 ]. Essendo uno dei principali percorsi di riparazione della rottura del DNA a doppio filamento (DSB), la riparazione dell'unione delle estremità non omologhe (NHEJ) svolge un ruolo critico nella ricombinazione V(D)J mediata dai linfociti specifici che attivano la ricombinazione genica (RAG), che si traduce in un repertorio molto diversificato di anticorpi nelle cellule B e recettori delle cellule T (TCR) nelle cellule T [ 8]. Ad esempio, la perdita di funzione delle proteine chiave di riparazione del DNA come ATM, DNA-PKcs, 53BP1, et al., porta a difetti nella riparazione NHEJ che inibiscono la produzione di cellule B e T funzionali, portando all'immunodeficienza [ 7 , 9 , 10 , 11 ]. Al contrario, l'infezione virale di solito induce danni al DNA attraverso meccanismi diversi, come l'induzione della produzione di specie reattive dell'ossigeno (ROS) e lo stress di replicazione delle cellule ospiti [ 12 , 13 , 14]. Se il danno al DNA non può essere adeguatamente riparato, contribuirà all'amplificazione della patologia indotta da infezione virale. Pertanto, abbiamo mirato a indagare se le proteine SARS-CoV-2 dirottano il sistema di riparazione del danno al DNA, influenzando così l'immunità adattativa in vitro.
2. Materiali e metodi
2.1. Anticorpi e reagenti
Gli anticorpi DAPI (Cat # MBD0015), doxorubicina (Cat # D1515), H 2 O 2 (Cat # H1009) e -tubulina (Cat # T4026) sono stati acquistati da Sigma-Aldrich. Anticorpi contro l'etichetta His (Cat #12698), H2A (Cat # 12349), H2A.X (Cat # 7631), γ–H2A.X (Cat # 2577), Ku80 (Cat # 2753) e Rad51 (Cat # 8875 ) sono stati acquistati da Cell Signaling Technology (Danvers, MA, USA). Gli anticorpi 53BP1 (Cat # NB100-304) e RNF168 (Cat # H00165918-M01) sono stati ottenuti da Novus Biologicals (Novus Biologicals, Littleton, CO, USA). Gli anticorpi Lamin B (Cat #sc–374015), ATM (Cat #sc–135663), DNA–PK (Cat #sc–5282) e BRCA1 (Cat #sc–28383) sono stati acquistati da Santa Cruz Biotechnology (Santa Cruz, CA, USA). L'anticorpo XRCC4 (Cat # PA5-82264) è stato acquistato da Thermo Fisher Scientific (Waltham, MA, USA).
2.2. plasmidi
pHPRT–DRGFP e pCBASceI sono stati gentilmente donati da Maria Jasin (plasmidi Addgene #26476 e #26477) [ 15 ]. pimEJ5GFP è stato un regalo di Jeremy Stark (plasmide Addgene #44026) [ 16]. Le proteine NSP1, NSP9, NSP13, NSP14, NSP16, spike e nucleocapside sono state prima sintetizzate con l'ottimizzazione del codone e quindi clonate in un vettore di espressione di mammifero pUC57 con un tag 6xHis C-terminale. Una sequenza complementare invertita RSS-GFP di 12 spaziatori, un RSS di 23 spaziatori è stata sintetizzata per il vettore reporter V(D)J. Quindi, la sequenza è stata clonata nel vettore pBabe-IRES-mRFP per generare il vettore reporter pBabe-12RSS-GFPi-23RSS-IRES-mRFP. Sequenza RSS 12–spacer: 5′–CACAGTGCTACAGACTGGAACAAAACC–3′. Sequenza RSS 23–spacer: 5′–CACAGTGGTAGTACTCCACTGTCTGGCTGTACAAAAAACC–3′. I costrutti di espressione RAG1 e RAG2 sono stati generosamente donati da Martin Gellert (plasmide Addgene #13328 e #13329) [ 17 ].
2.3. Cellule e coltura cellulare
Le cellule HEK293T e HEK293 ottenute dall'American Type Culture Collection (ATCC) sono state coltivate sotto il 5% di CO 2 a 37 ° C nel mezzo di Eagle modificato di Dulbecco (DMEM, high glucosio, GlutaMAX) (Life Technologies, Carlsbad, CA, USA) contenente 10 % ( v / v ) siero fetale di vitello (FCS, Gibco), 1% ( v / v ) penicillina (100 UI/mL) e streptomicina (100 μg/mL). Le cellule reporter HEK293T-DR-GFP e HEK293T-EJ5-GFP sono state generate come descritto in precedenza e coltivate sotto il 5% di CO 2 a 37 ° C nel suddetto mezzo di coltura.
2.4. Saggi per reporter HR e NHEJ
La riparazione di HR e NHEJ nelle cellule HEK293T è stata misurata come descritto in precedenza utilizzando cellule stabili DR-GFP e EJ5-GFP. In breve, 0,5 × 10 6 cellule reporter stabili HEK293T sono state seminate in piastre a 6 pozzetti e trasfettate con 2 μg di plasmide di espressione I-SceI (pCBASceI) insieme a plasmidi di espressione di proteine SARS-CoV-2. Quarantotto ore dopo la trasfezione e il trattamento con aspirina, le cellule sono state raccolte e analizzate mediante analisi di citometria a flusso per l'espressione di GFP. I mezzi sono stati ottenuti da tre esperimenti indipendenti.
2.5. Frazionamento cellulare e immunoblotting
Per il dosaggio della frazione cellulare, è stato utilizzato il kit di frazionamento delle proteine subcellulari (Thermo Fisher) secondo le istruzioni del produttore. I lisati proteici sono stati quantificati utilizzando il reagente BCA (Thermo Fisher Scientific, Rockford, IL, USA). Le proteine sono state risolte mediante elettroforesi su gel di sodio dodecil solfato-poliacrilammide (SDS-PAGE), trasferite su membrane di nitrocellulosa (Amersham protran, 0,45 μm NC) e immunoblotting con anticorpi primari specifici seguiti da anticorpi secondari coniugati con HRP. Le bande proteiche sono state rilevate utilizzando il kit SuperSignal West Pico o Femto Chemiluminescence (Thermo Fisher Scientific).
2.6. Comet Assay
Le cellule sono state trattate con diversi reagenti per il danno al DNA e quindi raccolte nei punti temporali indicati per l'analisi. Celle (1 × 10 5cellule/mL in soluzione salina tamponata con fosfato [PBS]) sono stati risospesi in agarosio a basso punto di fusione all'1% a 40 °C con un rapporto di 1:3 vol/vol e pipettati su un CometSlide. I vetrini sono stati quindi immersi in tampone di lisi preraffreddato (1,2 M NaCl, 100 mM EDTA, 0,1% sodio lauril sarcosinato, 0,26 M NaOH pH > 13) per la lisi durante la notte (18-20 h) a 4 ° C al buio. I vetrini sono stati quindi rimossi con cura e immersi in tampone di risciacquo (0,03 M NaOH e 2 mM EDTA, pH > 12) a temperatura ambiente (RT) per 20 minuti al buio. Questa fase di lavaggio è stata ripetuta due volte. I vetrini sono stati trasferiti in una camera di elettroforesi orizzontale contenente tampone di risciacquo e separati per 25 minuti ad una tensione di 0,6 V/cm. Infine, i vetrini sono stati lavati con acqua distillata, colorati con ioduro di propidio 10 μg/mL e analizzati mediante microscopia a fluorescenza.
2.7. Immunofluorescenza
Le cellule sono state seminate su vetrini coprioggetto in una piastra da 12 pozzetti e trasfettate con il plasmide indicato per 24 ore. Quindi, le cellule sono state trattate con o senza reagenti per il danno al DNA secondo il setup sperimentale. Le cellule sono state fissate in paraformaldeide (PFA) al 4% in PBS per 20 minuti a temperatura ambiente e quindi permeabilizzate in Triton X-100 allo 0,5% per 10 minuti. I vetrini sono stati bloccati in siero di capra normale al 5% (NGS) e incubati con anticorpi primari diluiti in NGS 1% durante la notte a 4 ° C. I campioni sono stati quindi incubati con gli anticorpi secondari indicati marcati con Alexa Fluor 488 o 555 (Invitrogen) diluiti in NGS all'1% a temperatura ambiente per 1 ora. Successivamente, sono stati colorati con DAPI per 15 minuti a temperatura ambiente. I coprioggetto sono stati montati utilizzando il mezzo di montaggio per fluorescenza Dako (Agilent) e ripreso con un microscopio confocale Nikon (Eclipse C1 Plus).
2.8. Analisi della ricombinazione V(D)J
In breve, il plasmide reporter V(D)J contiene GFP invertito e IRES che guidano RFP continuamente espressa. La RFP continuamente espressa è il controllo della trasfezione interna. Dopo che il gene di attivazione della ricombinazione 1/2 (RAG1/2) è stato co-trasfettato nelle cellule, RAG1/2 taglierà l'RSS e l'induzione mediata dei DSB, se si verifica la ricombinazione V(D)J, i GFP invertiti vengono ligati in ordine positivo da NHEJ riparazione. Quindi la cellula esprimerà GFP funzionale. Quindi, le cellule double positive GFP e RFP sono la lettura del test del reporter V(D)J [ 18]. Le cellule 293T al 70% di confluenza sono state trasfettate con il reporter V(D)J GFP da solo (sfondo) o in combinazione con i costrutti di espressione RAG1 e RAG2, con un rapporto di 1 µg Reporter V(D)J GFP: 0,5 µg RAG1: 0,5 µg RAG2. Il giorno seguente, il mezzo è stato cambiato e, dopo altre 48 ore, le cellule sono state raccolte e analizzate mediante citometria a flusso per l'espressione di GFP e RFP.
2.9. Analisi statistica
Tutti gli esperimenti sono stati ripetuti almeno tre volte utilizzando campioni raccolti o preparati indipendentemente. I dati sono stati analizzati mediante il test t di Student o ANOVA seguito dai test di confronto multiplo di Tukey utilizzando GraphPad 8.
3. Risultati
3.1. Effetto delle proteine virali SARS-CoV-2 localizzate nucleari sulla riparazione dei danni al DNA
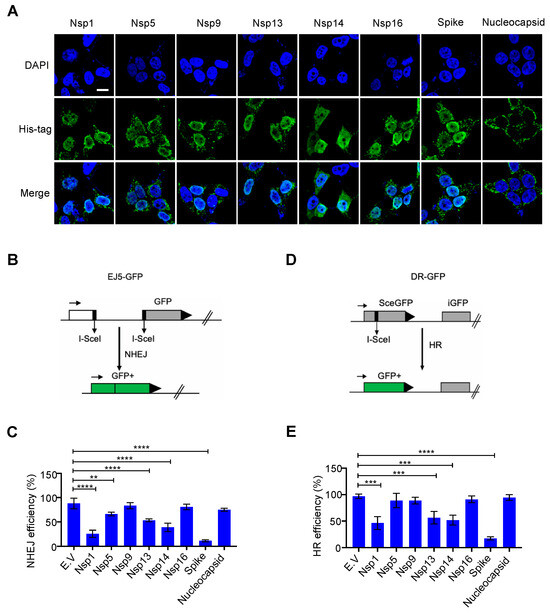
La riparazione del danno al DNA avviene principalmente nel nucleo per garantire la stabilità del genoma. Sebbene le proteine SARS-CoV-2 siano sintetizzate nel citosol [ 1 ], alcune proteine virali sono rilevabili anche nel nucleo, tra cui Nsp1, Nsp5, Nsp9, Nsp13, Nsp14 e Nsp16 [ 19 ]. Abbiamo studiato se queste proteine SARS-CoV-2 localizzate nel nucleo influenzino il sistema di riparazione dei danni al DNA della cellula ospite. Per questo, abbiamo costruito questi plasmidi di espressione di proteine virali insieme a plasmidi di espressione di spike e nucleoproteine, che sono generalmente considerate proteine localizzate nel citosol. Abbiamo confermato la loro espressione e localizzazione mediante immunoblotting e immunofluorescenza ( Figura 1 A e Figura S1A ). I nostri risultati erano coerenti con quelli di studi precedenti [19 ]; Le proteine Nsp1, Nsp5, Nsp9, Nsp13, Nsp14 e Nsp16 sono infatti localizzate nel nucleo e le nucleoproteine sono localizzate principalmente nel citosol. Sorprendentemente, abbiamo trovato l'abbondanza della proteina spike nel nucleo ( Figura 1 A). La riparazione NHEJ e la riparazione della ricombinazione omologa (HR) sono due principali percorsi di riparazione del DNA che non solo monitorano e garantiscono continuamente l'integrità del genoma, ma sono anche vitali per le funzioni adattative delle cellule immunitarie [ 9 ]. Per valutare se queste proteine virali ostacolano la via di riparazione del DSB, abbiamo esaminato la riparazione di un DSB sito-specifico indotto dall'endonucleasi I-SceI utilizzando la proteina fluorescente verde a ripetizione diretta (DR-GFP) e la NHEJ-GFP totale ( EJ5-GFP) sistemi reporter per HR e NHEJ, rispettivamente [ 15 , 16]. La sovraespressione di Nsp1, Nsp5, Nsp13, Nsp14 e le proteine spike hanno diminuito l'efficienza della riparazione sia di HR che di NHEJ ( Figura 1 B–E e Figura S2A,B ). Inoltre, abbiamo anche scoperto che la sovraespressione di Nsp1, Nsp5, Nsp13 e Nsp14 sopprimeva drasticamente la proliferazione rispetto ad altre proteine studiate ( Figura S3A,B ). Pertanto, l'effetto inibitorio di Nsp1, Nsp5, Nsp13 e Nsp14 sulla riparazione del danno al DNA può essere dovuto a effetti secondari, come l'arresto della crescita e la morte cellulare. È interessante notare che la proteina spike sovraespressa non ha influenzato la morfologia o la proliferazione cellulare, ma ha soppresso significativamente sia la riparazione HR che NHEJ ( Figura 1 B-E, Figure S2A, B e S3A, B ).
Figura 1. Effetto delle proteine localizzate nel nucleo del coronavirus 2 (SARS-CoV-2) della sindrome respiratoria acuta grave sulla riparazione del danno al DNA. ( A ) Distribuzione subcellulare delle proteine SARS-CoV-2. L'immunofluorescenza è stata eseguita 24 ore dopo la trasfezione del plasmide che esprime le proteine virali nelle cellule HEK293T. Barra della scala: 10 µm. ( B ) Schema del reporter EJ5-GFP utilizzato per monitorare l'unione delle estremità non omologhe (NHEJ). ( C ) Effetto del vettore vuoto (EV) e delle proteine SARS-CoV-2 sulla riparazione del DNA NHEJ. I valori rappresentano la media ± deviazione standard (DS) da tre esperimenti indipendenti (vedi grafici FACS rappresentativi nella Figura S2A ). ( D) Schema del reporter DR-GFP utilizzato per monitorare la ricombinazione omologa (HR). ( E ) Effetto delle proteine EV e SARS-CoV-2 sulla riparazione del DNA HR. I valori rappresentano la media ± SD da tre esperimenti indipendenti (vedi grafici FACS rappresentativi nella Figura S2B ). I valori rappresentano la media ± DS, n = 3. La significatività statistica è stata determinata utilizzando l'analisi della varianza unidirezionale (ANOVA) in ( C , E ). ** p < 0,01, *** p < 0,001, **** p < 0,0001.
3.2. La proteina Spike SARS-CoV-2 inibisce la riparazione dei danni al DNA
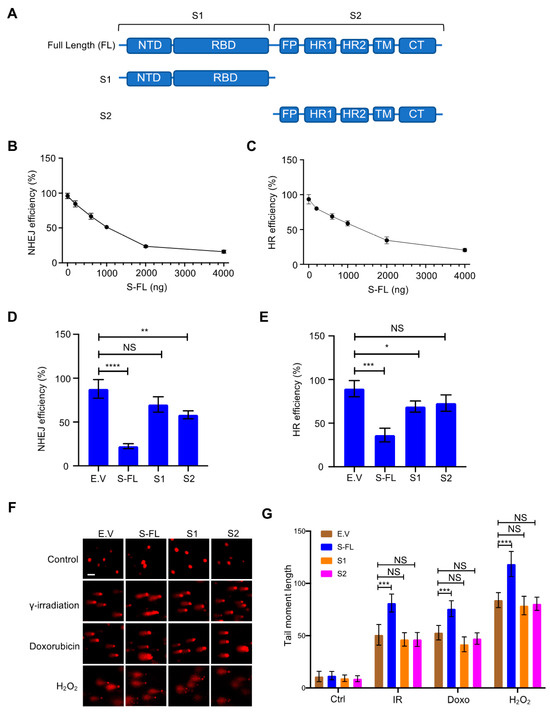
Poiché le proteine spike sono fondamentali per mediare l'ingresso virale nelle cellule ospiti e sono al centro della maggior parte delle strategie vaccinali [ 20 , 21 ], abbiamo ulteriormente studiato il ruolo delle proteine spike nella riparazione del danno al DNA e nella sua ricombinazione V(D)J associata. Di solito si pensa che le proteine Spike siano sintetizzate sul reticolo endoplasmatico ruvido (ER) [ 1 ]. Dopo modifiche post-traduzionali come la glicosilazione, le proteine spike transitano attraverso l'apparato della membrana cellulare insieme ad altre proteine virali per formare il virione maturo [ 1 ]. La proteina Spike contiene due subunità principali, S1 e S2, oltre a diversi domini funzionali o ripetizioni [ 22 ] ( Figura 2UN). Nello stato nativo, le proteine spike esistono come proteine intere inattive. Durante l'infezione virale, le proteasi della cellula ospite come la furin proteasi attivano la proteina S scindendola nelle subunità S1 e S2, che è necessaria per l'ingresso del virus nella cellula bersaglio [ 23 ]. Abbiamo ulteriormente esplorato diverse subunità della proteina spike per chiarire le caratteristiche funzionali richieste per l'inibizione della riparazione del DNA. Solo la proteina spike a lunghezza intera ha fortemente inibito sia la riparazione NHEJ che HR ( Figura 2 B-E e Figura S4A, B). Successivamente, abbiamo cercato di determinare se la proteina spike contribuisce direttamente all'instabilità genomica inibendo la riparazione del DSB. Abbiamo monitorato i livelli di DSB utilizzando comet assay. A seguito di diversi trattamenti per il danno al DNA, come l'irradiazione , il trattamento con doxorubicina e il trattamento con H 2 O 2 , c'è meno riparazione in presenza della proteina spike ( Figura 2 F, G). Insieme, questi dati dimostrano che la proteina spike influenza direttamente la riparazione del DNA nel nucleo.
Figura 2. La proteina spike della sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) inibisce la riparazione del danno al DNA. ( A ) Schema della struttura primaria della proteina spike SARS-CoV-2. La subunità S1 include un dominio N-terminale (NTD, 14–305 residui) e un dominio legante il recettore (RBD, 319–541 residui). La subunità S2 è costituita dal peptide di fusione (FP, 788-806 residui), sequenza di ripetizione dell'eptapeptide 1 (HR1, 912-984 residui), HR2 (1163-1213 residui), dominio TM (TM, 1213-1237 residui) e dominio del citoplasma (CT,1237-1273 residui). ( B , C ) Effetto dell'espressione titolata della proteina spike sulla riparazione del DNA nelle cellule HEK-293T. ( D , E) Solo la proteina spike a lunghezza intera inibisce la riparazione del DNA di giunzione non omologa (NHEJ) e di ricombinazione omologa (HR). I valori rappresentano la media ± SD da tre esperimenti indipendenti (vedi grafici FACS rappresentativi nella Figura S4A,B ). ( F ) Le cellule HEK293T trasfettate con proteina spike a lunghezza intera (S-FL) hanno mostrato più danni al DNA rispetto alle cellule trasfettate con vettore vuoto, S1 e S2 in diverse condizioni di danno al DNA. Per la doxorubicina: 4 µg/mL, 2 h. Per irradiazione : 10 Gy, 30 min. Per H 2 O 2 : 100 µM, 1 h. Barra della scala: 50 µm. ( G ) Quantificazione corrispondente dei momenti della coda della cometa da 20 diversi campi con n> 200 comete di tre esperimenti indipendenti. La significatività statistica è stata valutata utilizzando un'analisi della varianza a due vie (ANOVA). NS (non significativo): * p > 0,05, ** p < 0,01, *** p < 0,001, **** p < 0,0001.
3.3. Le proteine spike impediscono il reclutamento di proteine del checkpoint per la riparazione del danno al DNA
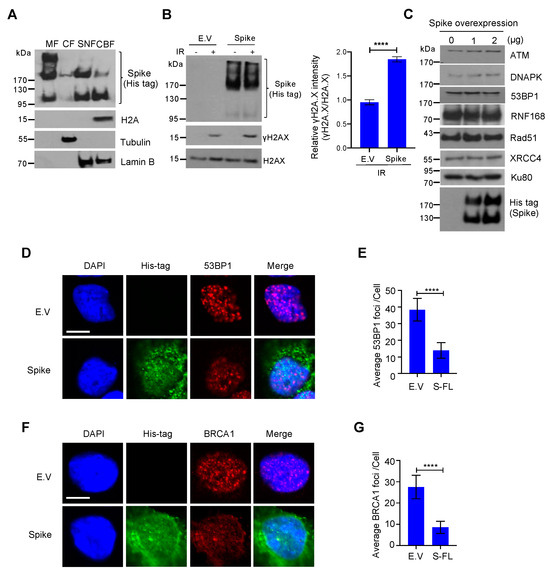
Per confermare l'esistenza della proteina spike nel nucleo, abbiamo eseguito l'analisi della frazione subcellulare e abbiamo scoperto che le proteine spike non sono solo arricchite nella frazione di membrana cellulare, ma sono anche abbondanti nella frazione nucleare, con espressione rilevabile anche nella frazione legata alla cromatina. Figura 3A). Abbiamo anche osservato che il picco ha tre forme diverse, la banda superiore è un picco altamente glicosilato, quello centrale è un picco a tutta lunghezza e quello inferiore è una subunità di picco scissa. Coerentemente con il test della cometa, abbiamo anche trovato la sovraregolazione del marcatore di danno al DNA, -H2A.X, in cellule sovraespresse di proteine spike in condizioni di danno al DNA ( Figura 3 B). Uno studio recente ha suggerito che le proteine spike inducono stress ER e degradazione proteica associata a ER [24 ]. Per escludere la possibilità che la proteina spike inibisca la riparazione del DNA promuovendo la degradazione della proteina di riparazione del DNA, abbiamo controllato l'espressione di alcune proteine essenziali di riparazione del DNA nei percorsi di riparazione NHEJ e HR e abbiamo scoperto che queste proteine di riparazione del DNA erano stabili dopo la sovraespressione della proteina spike ( Figura 3 C). Per determinare come la proteina spike inibisce i percorsi di riparazione sia NHEJ che HR, abbiamo analizzato il reclutamento di BRCA1 e 53BP1, che sono le proteine chiave del checkpoint per la riparazione HR e NHEJ, rispettivamente. Abbiamo scoperto che la proteina spike ha inibito notevolmente la formazione di focolai sia BRCA1 che 53BP1 ( Figura 3 D–G). Insieme, questi dati mostrano che la proteina spike a lunghezza intera SARS-CoV-2 inibisce la riparazione del danno al DNA ostacolando il reclutamento della proteina di riparazione del DNA.
Figura
3. La proteina spike della sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) impedisce il reclutamento di proteine del punto di controllo per la riparazione del danno al DNA. ( A ) La frazione di membrana (MF), la frazione citosolica (CF), la frazione nucleare solubile (SNF) e la frazione legata alla cromatina (CBF) da cellule HEK293T trasfettate con la proteina spike SARS-CoV-2 sono state immunoblotting per il picco His-tag e proteine indicate. ( B ) A sinistra: immunoblot del marcatore di danno al DNA γH2AX nel vettore vuoto (EV) e cellule HEK293T che esprimono proteine spike dopo 10 Gy di irradiazione . A destra: quantificazione corrispondente di immunoblot a sinistra. I valori rappresentano la media ± SD ( n = 3). La significatività statistica è stata determinata utilizzando il test t di Student . **** p < 0,0001. ( C ) Immunoblots di proteine correlate alla riparazione del danno al DNA in cellule HEK293T che esprimono proteine spike. ( D ) Immagini rappresentative della formazione di focolai 53BP1 in cellule HEK293 che esprimono proteine EV e spike esposte a 10 Gy -irradiazione. Barra della scala: 10 µm. ( E ) Analisi quantitativa dei fuochi 53BP1 per nucleo. I valori rappresentano la media ± SEM, n = 50. ( F ) Formazione di focolai BRCA1 in cellule HEK293 che esprimono proteine vector e spike vuote esposte a 10 Gy -irradiazione. Barra della scala: 10 µm. ( G ). Analisi quantitativa dei fuochi BRCA1 per nucleo. I valori rappresentano la media ± SEM, n = 50. La significatività statistica è stata determinata utilizzando t . di Student-test. **** p < 0,0001.3.4. La proteina Spike altera la ricombinazione V(D)J In vitro
La riparazione del danno al DNA, in particolare la riparazione NHEJ, è essenziale per la ricombinazione V(D)J, che si trova al centro dell'immunità delle cellule B e T [ 9 ]. Ad oggi, molti vaccini SARS-CoV-2 approvati, come i vaccini mRNA e i vaccini adenovirus-COVID-19, sono stati sviluppati sulla base della proteina spike a lunghezza intera [ 25 ]. Sebbene sia discutibile se SARS-CoV-2 infetti direttamente i precursori dei linfociti [ 26 , 27 ], alcuni rapporti hanno dimostrato che le cellule infette secernono esosomi in grado di fornire RNA o proteine SARS-CoV-2 alle cellule bersaglio [ 28 , 29]. Abbiamo ulteriormente testato se la proteina spike riducesse la ricombinazione V(D)J mediata da NHEJ. Per questo, abbiamo progettato un sistema reporter di ricombinazione V(D)J in vitro secondo uno studio precedente [ 18 ] ( Figura S5 ). Rispetto al vettore vuoto, la sovraespressione della proteina spike ha inibito la ricombinazione V(D)J mediata da RAG in questo sistema reporter in vitro ( Figura 4 ).
Figura 4. La proteina Spike altera la ricombinazione V(D)J in vitro. ( A ) Schema del sistema reporter V(D)J. ( B ) I grafici rappresentativi della citometria a flusso mostrano che la proteina spike SARS-CoV-2 impedisce la ricombinazione V (D) J in vitro. ( C ) Analisi quantitativa della ricombinazione relativa V(D)J. I valori rappresentano la media ± DS, n = 3. La significatività statistica è stata determinata utilizzando il test t di Student . **** p < 0,0001.
4. Discussione
I nostri risultati forniscono prove della proteina spike che dirotta il meccanismo di riparazione del danno al DNA e il meccanismo immunitario adattativo in vitro. Proponiamo un potenziale meccanismo mediante il quale le proteine spike possono compromettere l'immunità adattativa inibendo la riparazione del danno al DNA. Sebbene non sia stata pubblicata alcuna prova che SARS-CoV-2 possa infettare i timociti o le cellule linfoidi del midollo osseo, il nostro test del reporter V(D)J in vitro mostra che la proteina spike ha intensamente impedito la ricombinazione V(D)J. Coerentemente con i nostri risultati, le osservazioni cliniche mostrano anche che il rischio di malattia grave o morte con COVID-19 aumenta con l'età, in particolare gli anziani che sono a più alto rischio [ 22]. Ciò può essere dovuto al fatto che le proteine spike di SARS-CoV-2 possono indebolire il sistema di riparazione del DNA delle persone anziane e di conseguenza impedire la ricombinazione V(D)J e l'immunità adattativa. Al contrario, i nostri dati forniscono dettagli preziosi sul coinvolgimento delle subunità proteiche spike nella riparazione del danno al DNA, indicando che i vaccini basati su spike a lunghezza intera possono inibire la ricombinazione di V(D)J nelle cellule B, il che è anche coerente con un recente studio che un vaccino a lunghezza intera basato su spike ha indotto titoli anticorpali inferiori rispetto al vaccino a base di RBD [ 28]. Ciò suggerisce che l'uso di epitopi antigenici del picco come vaccino SARS-CoV-2 potrebbe essere più sicuro ed efficace del picco a lunghezza intera. Nel loro insieme, abbiamo identificato uno dei meccanismi potenzialmente importanti della soppressione di SARS-CoV-2 del meccanismo immunitario adattativo dell'ospite. Inoltre, i nostri risultati implicano anche un potenziale effetto collaterale del vaccino a lunghezza intera basato su spike. Questo lavoro migliorerà la comprensione della patogenesi del COVID-19 e fornirà nuove strategie per la progettazione di vaccini più efficienti e più sicuri.
Materiali supplementari
I seguenti sono disponibili online all'indirizzo https://www.mdpi.com/article/10.3390/v13102056/s1






Ovvero:
RispondiEliminaIl "vaccino", sopprimendo il meccanismo naturale di riparazione del DNA rende le persone altamente suscettibili a mutazioni cancerose anche se esposte a livelli molto bassi di radiazioni ionizzanti come l'esposizione alla luce solare o la mammografia etc...